Arbeitsgruppe Dr. Daniel Maag
Dr. Daniel Maag
Lehrstuhl Pharmazeutische Biologie
Julius-von-Sachs-Institut für Biowissenschaften
Julius-von-Sachs-Platz 2
97082 Würzburg
Tel.: + 49 931 31-85538
Fax: + 49 931 31-86182
daniel.maag@uni-wuerzburg.de
CV
Publikationen
Forschungsschwerpunkt
In ihrem natürlichen Lebensraum sind die meisten Pflanzen mit ständig wechselnden Temperaturen konfrontiert, die ihren Fortpflanzungserfolg bedrohen können. Pflanzen sind jedoch in der Lage, diese Temperaturänderungen wahrzunehmen und ihr Wachstum und ihre Entwicklung entsprechend anzupassen. In meiner Gruppe untersuchen wir verschiedene Aspekte der pflanzlichen Reaktionen auf erhöhte Temperaturen, die von morphologischen Anpassungen im Laufe mehrerer Tage bis hin zu sehr schnellen Reaktionen auf molekularer Ebene reichen, anhand des Modellorganismus Arabidopsis thaliana. Letztlich zielt unsere Forschung auf ein besseres Verständnis der Temperaturwahrnehmung von Pflanzen sowie der zugrundeliegenden regulatorischen Netzwerke ab, was eine Voraussetzung für die Züchtung klimaresistenter Pflanzensorten darstellt.

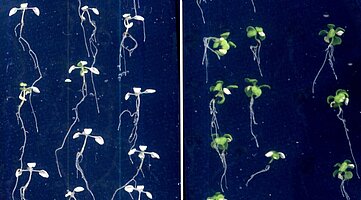
Untersuchung der frühen Signalwege während der Thermomorphogenese
Bei moderatem Hitzestress zeigen Arabidopsis-Keimlinge ein Wachstumssyndrom, das als Thermomorphogenese zusammengefasst wird und zur Kühlung der Blätter durch eine erhöhte Transpirationsrate beiträgt. Zu diesen morphologischen Anpassungen zählt beispielsweise das verstärkte Streckungswachstum des Hypokotyls und der Blattstiele. Innerhalb dieses Projektes zielen wir auf die Identifizierung molekularer Regulatoren ab, die an der frühen Signalübertragung während der thermomorphogenetischen Wachstumsanpassungen beteiligt sind.
Identifikation neuer Regulatoren der pflanzlichen Hitzeschockantwort
Gemäß jüngeren wissenschaftlichen Studien zeigt Arabidopsis thaliana eine beachtliche intraspezifische Variabilität in ihrer Temperaturantwort. Im Rahmen dieses Projekts machen wir uns diese intraspezifische Variabilität zunutze, um neue Regulatoren der pflanzlichen Hitzeschockantwort zu identifizieren. Hierfür greifen wir auf eine Kollektion von über 1000 A. thaliana Linien zurück, deren Genome im Jahr 2016 durch das 1001 Genomes Consortium sequenziert und veröffentlicht wurden. Die Herkunftsorte dieser Linien umspannen das gesamte natürliche Verbreitungsgebiet von A. thaliana und stellen eine ideale Ressource für die Durchführung von genomweiten Assoziationsstudien (GWAS) zur Untersuchung der genetischen Grundlagen der erworbenen Thermotoleranz dar. Die Umsetzung dieses Projektes erfolgt in enger Zusammenarbeit mit der Gruppe von Arthur Korte am Centre for Computational and Theoretical Biology (CCTB) der Universität Würzburg.