Software Toolbox
R Packages auf Bioconductor
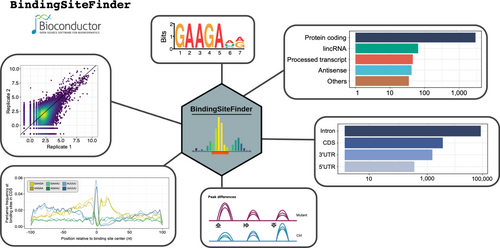
Das R/Bioconductor Package BindingSiteFinder bietet einen vollständigen Arbeitsablauf für die Definition der Bindestellen eines RNA-bindenden Proteins (RBPs) auf Basis von iCLIP- und eCLIP-Daten. Das Package bietet sowohl Funktionen als auch reichhaltige Visualisierungen und ist gut in moderne und weit verbreitete Bioconductor-Klassen wie GenomicRanges und SummarizedExperiments integriert. Das DifferentialBinding-Modul erweitert den BindingSiteFinder um verschiedene neue Funktionen, die geeignet sind, RBP-spezifische Bindestellenveränderungen transkriptomweit zu identifizieren.
Die Verwendung wird in der Vignette im Detail erklärt.
Eine einfache und schnelle Möglichkeit zur Visualisierung und Profilierung von Hochdurchsatz-IP-Daten. Dieses Paket erzeugt das Meta-Genprofil und andere Profile. Diese Profile können wertvolle Informationen zum Verständnis der IP-Experimentergebnisse liefern.
Die Verwendung wird in der Vignette im Detail erklärt.
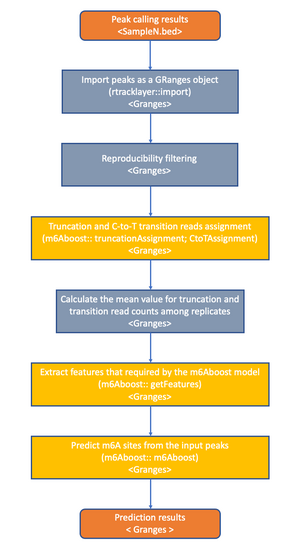
Dieses Paket kann dem Benutzer helfen, das m6Aboost-Modell auf seinen eigenen miCLIP2-Daten laufen zu lassen. Das Paket enthält Funktionen zum Zuweisen des Siganls sowie um die features des m6Aboost-Modells zu erhalten und zu verwenden.
m6A individual-nucleotide resolution UV crosslinking and immunoprecipitation (miCLIP) und das verbesserte miCLIP2 sind m6A-Antikörper-basierte Methoden, die die transkriptomweite Kartierung von m6A-Sites mit Einzel-Nukleotid-Auflösung ermöglichen (Körtel et al. 2021) (Linder et al. 2015). Aufgrund der begrenzten Spezifität und der hohen Kreuzreaktivität der m6A-Antikörper weisen die miCLIP-Daten jedoch ein hohes Hintergrundsignal auf, was die zuverlässige Identifizierung von m6A-Stellen anhand der Daten erschwert.
Zur genauen Erkennung von m6A-Stellen aus miCLIP- oder miCLIP2-Daten implementierten wir ein AdaBoost-basiertes maschinelles Lernmodell (m6Aboost) zur Klassifizierung der miCLIP2-Peaks in m6A-Stellen und Hintergrundsignale (Körtel et al. 2021). Das Modell wurde auf m6A-Stellen mit hoher Zuverlässigkeit trainiert, die durch den Vergleich von Wildtyp- und Mettl3-Knockout-Mausembryonalstammzellen, denen die Hauptmethyltransferase Mettl3 fehlt, gewonnen wurden. Für die Klassifizierung verwendet das m6Aboost-Modell eine Reihe von Merkmalen, darunter das experimentelle miCLIP2-Signal (Abbruchereignisse und C-zu-T-Übergänge) sowie die Transkriptregion (5'UTR, CDS, 3'UTR) und die Nukleotidsequenz in einem 21-NT-Fenster um den miCLIP2-Peak.
Die Verwendung wird in der Vignette im Detail erklärt.
Commandline tools
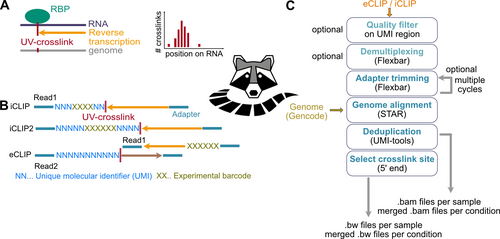
Workflow zur Analyse von CLIP Daten
Die genaue Kenntnis der Bindungsstellen eines RNA-bindenden Proteins (RBP) ist der Schlüssel zum Verständnis der (post-)transkriptionellen Genregulation. Diese Informationen können aus UV-Vernetzungs- und Immunpräzipitations-Experimenten (iCLIP) mit Einzel-Nukleotid-Auflösung gewonnen werden. In Busch et al 2020 haben wir den kompletten bioinformatischen Datenanalyse-Workflow beschrieben, um RBP-Bindungsstellen aus iCLIP-Daten zuverlässig zu erkennen. Der Arbeitsablauf umfasst alle Schritte von der anfänglichen Qualitätskontrolle der Sequenzierdaten bis zum Peak-Calling und der Quantifizierung der RBP-Bindung. Für jedes Tool werden die spezifischen Anforderungen für die iCLIP-Datenanalyse erläutert und optimierte Parametereinstellungen vorgeschlagen. Der Teil der Bindestellendefinition ist im BindingSiteFinderPackage implementiert (Mehr Informationen zu BindingSiteFinder hier).
Vereinfachte Nutzung des Workflows mit racoon_clip
Um die Verwendung des Workflows zu vereinfachen haben wir racoon_clip und BindingSiteFinder implementiert. racoon_clip ist ein coomandline tool dass die Prozessierung von CLIP Daten automatisiert (Klostermann & Zarnack 2024). Es kann sowohl iCLIP- als auch eCLIP-Daten verarbeiten und gibt das Crosslink-Signal mit Einzel-Nukleotid-Auflösung aus.
Die Nutzung von racoon_clip ist in der Dokumantation erklärt.
Detektion und Analyse von circRNAs mit Calcifer
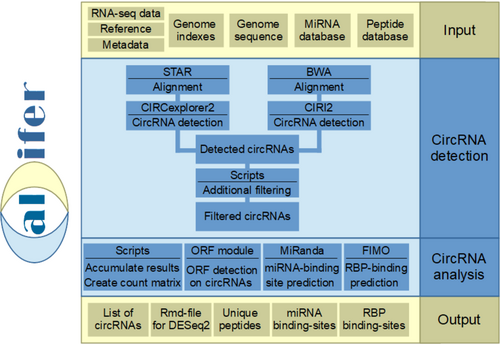
Calcifer ist ein Workflow für die hochautomatisierte Erkennung und Analyse von circRNAs in RNA-Seq-Datensätzen. Er ermöglicht die Auswertung von RNA-Seq Daten zur Identifikation Liste von charakterisierten circRNA-Isoformen sowie die Vorhersage möglicher Funktionen. Dabei werden zunächste die circRNAs aus den Daten detektierert und gefiltert. Dann wird die Anzahl der Reads auf den circRNAs und lineraren RNAs gezählt. Zum Schluss werden in den circRNAs miRNA binding sites, RBP binding sites und offene Leserahmen gesucht.
In Brezski et al 2024 wurde Calcifer verwendet um circRNAs in verschiedenen subnuclearen Kompartimenten zu vergleichen.
Alle Skripte zum Workflow sind im Calcifier GitHub repository zu finden.