RNA-Modifikationen
N6-Methyladenosin (m6A) ist die vorherrschende interne RNA-Modifikation in eukaryotischen Boten-RNAs (mRNAs) und spielt eine entscheidende Rolle für die mRNA-Stabilität.
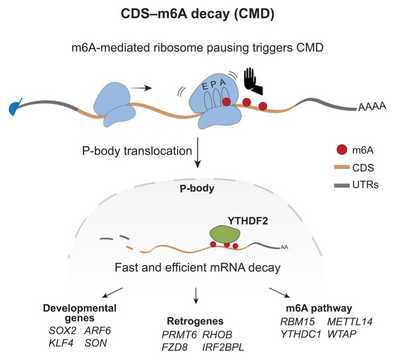
In diesem Projekt zeigen wir anhand menschlicher Zellen, dass m6A-Stellen in der kodierenden Sequenz (CDS) den CDS-m6A-Zerfall (CMD) auslösen, einen Weg, der sich von den bisher berichteten m6A-abhängigen Abbaumechanismen unterscheidet. Wichtig ist, dass die m6A-Stellen in der CDS wesentlich schneller und effizienter wirken als die in der 3′ untranslatierten Region, die bisher als Haupteffektoren angesehen wurden. Mechanistisch gesehen hängt CMD von der Translation ab, wobei die Ablagerung von m6A im CDS eine Ribosomenpause und die Destabilisierung des Transkripts auslöst. Der anschließende Zerfall beinhaltet die Translokation der CMD-Zieltranskripte in Prozessierungskörper (P-Körper) und die Rekrutierung des m6A-Leseproteins YT521-B homology domain family protein 2 (YTHDF2). Unsere Ergebnisse zeigen, dass CMD ein bisher unbekannter Weg ist, der besonders wichtig für die Kontrolle der Expression von Entwicklungsregulatoren und Retrogenen ist.
Dieses Projekt wurde in Zhou et al. 2024 publiziert.